
发表期刊:Cell Death Dis
影响因子:9.685
发表时间:2021年8月9日
研究方法:m5C bis-seq、CO-IP、qRT-PCR
文章链接:NSUN2 modified by SUMO-2/3 promotes gastric cancer progression and regulates mRNA m5C methylation
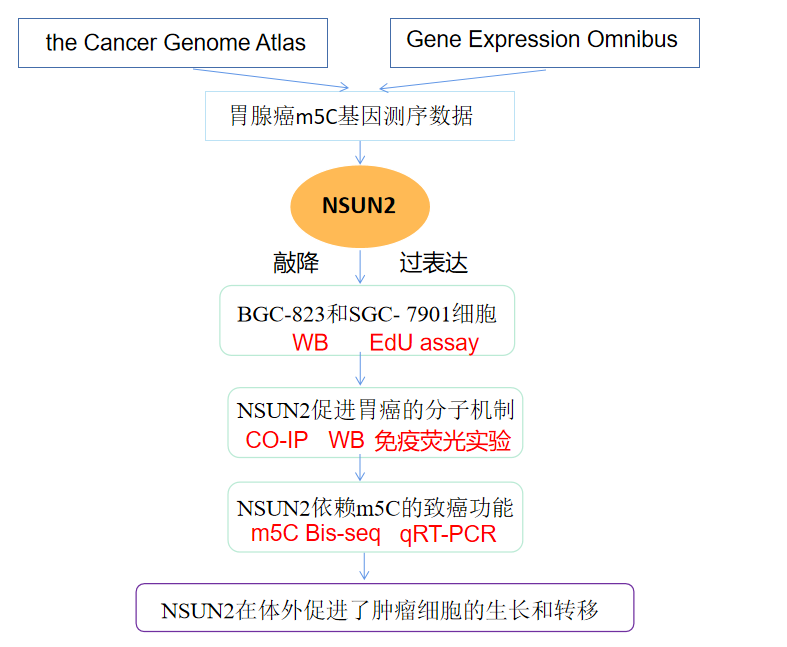
1.GC中m5C调控因子的表达景观及临床相关性
为了评估m5C调控因子在GC中的表达谱,作者从TCGA胃腺癌(STAD)项目中提取了与m5C相关的基因测序数据。在热图中展示了基因的整体表达水平(图1A)。与对照组相比,m5C调节因子在GC样品中普遍高表达,并且通过m5C调节网络描述了它们的相互作用和对胃癌患者总生存率的影响(图1B)。结果表明,NSUN2与其他m5C调控因子密切相关,且m5C调控因子的整体表达水平与患者的总生存率显著相关。
2.NSUN2在胃癌中高表达,并与不良预后相关
根据TCGA数据,作者发现NSUN2在多种癌症中均过表达(图1C)。为了进一步研究NSUN2的表达模式和胃癌患者的临床意义,作者用GC组织芯片对NSUN2进行了IHC染色,IHC结果显示,NSUN2主要在癌细胞的细胞核中表达,部分在细胞质中表达(图1D)。NSUN2在肿瘤组织中的表达高于在邻近正常组织中的肿瘤组织(图1E-F)。此外,Kaplan-Meier生存分析结果也表明,NSUN2高表达的胃癌患者总生存期(OS)低于NSUN2低表达的胃癌患者(图1G)。
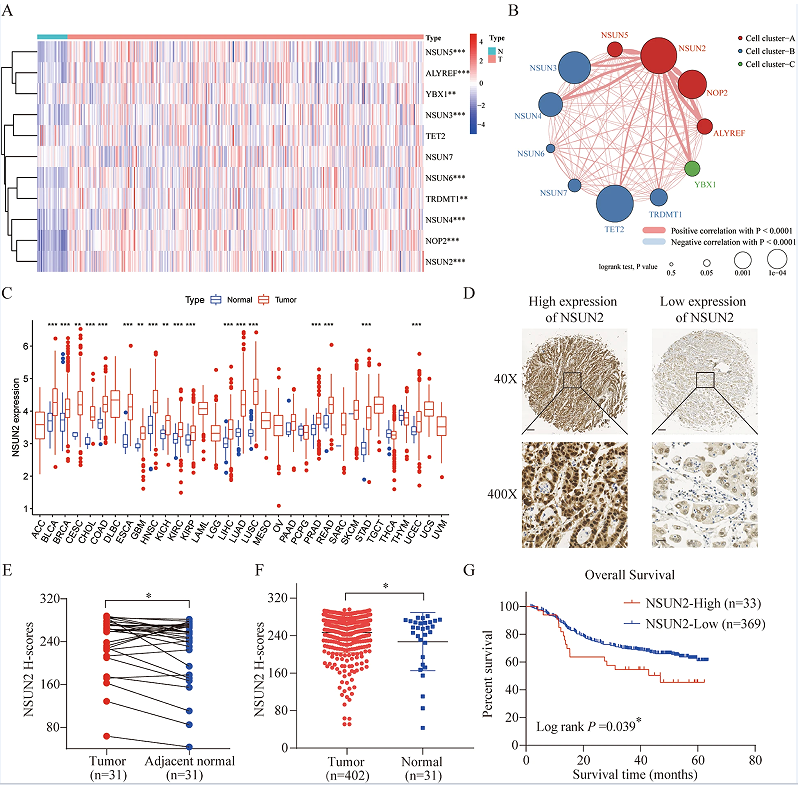
图1.RNAm5C调控因子在胃癌(GC)和NSUN2中的表达景观及临床相关性
3.NSUN2可促进胃癌细胞的增殖和转移
为了研究NSUN2在GC细胞中的致瘤作用,作者用siRNA敲除BGC-823和SGC- 7901细胞中的NSUN2。通过免疫印迹法检测,发现NSUN2的表达显著降低(图2A)。之后,为了确定NSUN2对GC细胞增殖的调控作用,作者进行了CCK-8和5-乙基-2’-脱氧尿苷(EdU)检测,发现NSUN2的下调导致BGC-823和SGC-7901细胞的增殖率下降(图2B、D)。与对照组相比,NSUN2过表达导致细胞增殖率显著增加(图2E-I)。采用集落形成试验来确定NUSN2对GC细胞增殖的长期影响。结果发现,两周后,NSUN2敲除组形成的菌落较少,而与对照组相比,NSUN2过表达组形成的菌落数量有所增加(图2C、H)。综上所述,NSUN2可促进GC细胞的增殖。
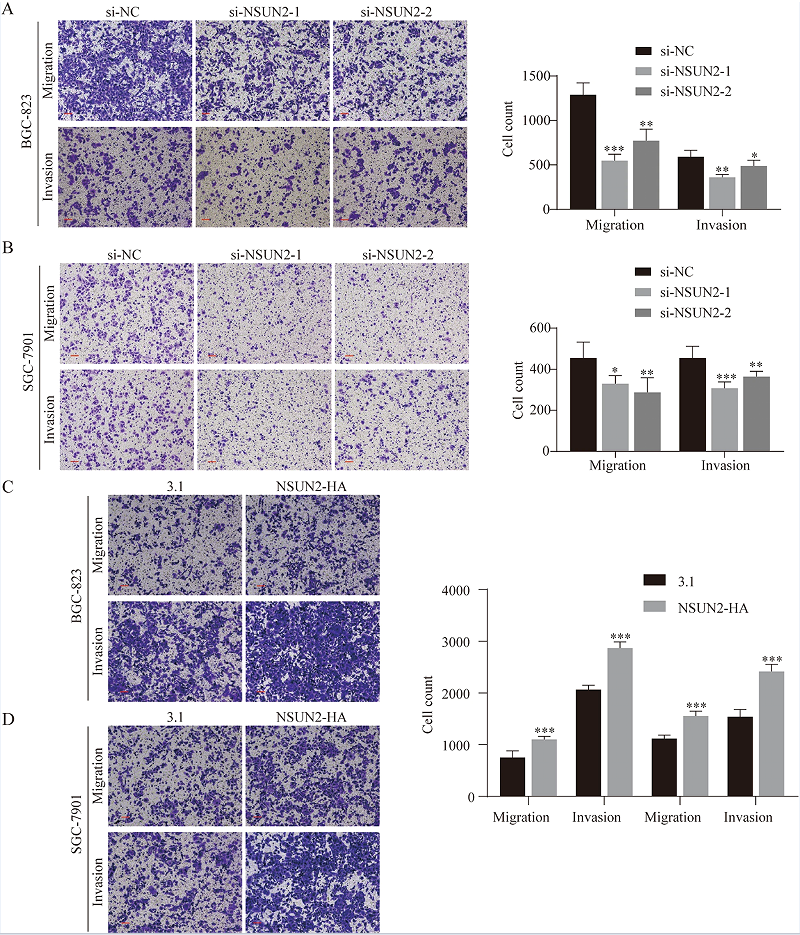
为了进一步探索NSUN2在GC进展中的作用,作者在BGC-823和SGC-7901细胞中进行了NSUN2敲低和过表达的迁移和侵袭实验。结果显示,与si-NC组相比,转染了si-NSUN2的GC细胞的迁移和入侵细胞数量显著减少(图3A-B),与对照组相比,过表达NSUN2的GC细胞的迁移和侵袭能力有所增加(图3C-D)。
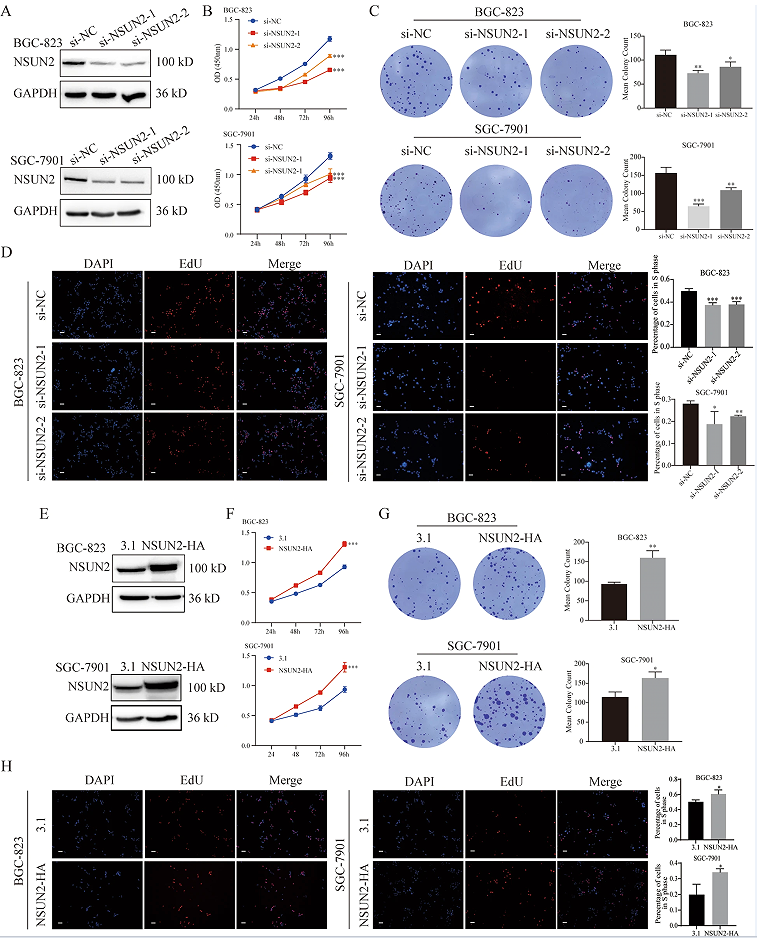
图2 NSUN2促进人胃癌(GC)细胞的生长
4.SUMO-2/3调节NSUN2的蛋白稳定性和亚细胞定位
为了探讨NSUN2促进GC的分子机制,作者通过进行免疫共沉淀(CO-IP)和质谱分析,研究了蛋白与NSUN2相互作用。CO-IP结果显示,SUMO-2/3被NSUN2-HA拉下,表现出直接的相互作用(图4A),并且还发现了两个SUMO相互作用基序(SIMs)和五个SUMOylation共识位点(图4B)。之后,通过两个突变质粒的构建,作者将NSUN2- HA、Δ236-240aa和Δ497-711aa转染到BGC-823细胞中,并进行CO-IP检测。发现转染NSUN2-Δ236-240aa后,NSUN2与SUMO-2/3之间的相互作用几乎消失,而转染NSUN2-Δ497-711aa后的相互作用略有下降(图4C)。此外,为了探讨NSUN2的SUMO化修饰是否影响蛋白水平。作者将抗SUMO-2/3的si-rna转染到BGC-823细胞中,并通过蛋白印迹法检测NSUN2蛋白水平,结果表明在SUMO-2/3基因敲除后,NSUN2的总蛋白水平几乎没有下降(图4D)。为了研究SUMO化修饰是否能改变NSUN2的亚细胞定位,作者转染了NSUN2-HA质粒,发现过表达NSUN2导致NSUN2在细胞质和细胞核中的表达显著增加(图4E)。将NSUN2-HA和SUMO-2/3si-RNAs共转染到细胞中,并分离出细胞质和核蛋白。结果显示在SUMO-2/3基因敲低后,NSUN2蛋白在细胞质中的水平略有下降,而在细胞核中的水平显著下降(图4F)。对NSUN2敲除的MGC-803(图4G)和BGC-823进行免疫荧光(IF)检测。检测NSUN2-HA和si-SUMO-3-2转染后NSUN2的亚细胞分布,结果表明SUMO-2/3基因敲低抑制了NSUN2向细胞核的易位。
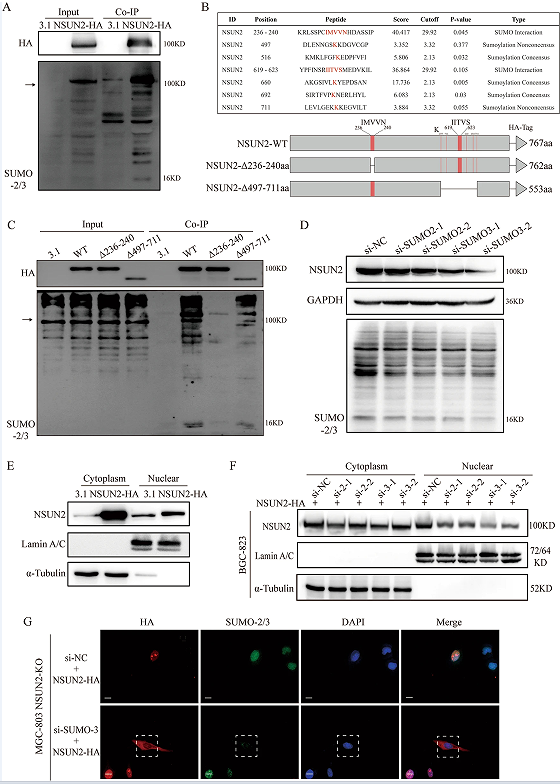
图4 SUMO-2/3与NSUN2相互作用,保持其稳定性,促进其核易位
5.NSUN2通过m5C依赖和独立机制促进胃进展
为了确定NSUN2的致癌功能是否依赖于其m5C甲基转移酶活性,作者通过引入释放(半胱氨酸271)和催化(半胱氨酸321)位点的点突变,生成了NSUN2的两个酶死亡突变体(图5A)。通过过表达NSUN2野生型和突变质粒BGC-823NSUN2-prhinty细胞系,发现NSUN2的野生型和酶死突变体能够部分拯救GC细胞的增殖和转移的能力,和野生型NSUN2呈现更重要的功能(图5B-C),这些结果表明,NSUN2通过m5C依赖和独立的机制促进胃进展。为了探索NSUN2依赖于m5C的功能,作者用m5C Bis-Seq获得了GC细胞中NSUN2敲除后转录组范围内的m5C修饰。对WT在NSUN2敲除细胞中分别鉴定出3260和712个m5C峰和135和70个独特基因(图5D),证实了真核mRNA中的m5C修饰主要是由NSUN2催化的。并且根据RNA转录本中m5C峰的定位,将其分为5’UTR区、StartC、编码序列(CDS)、StopC和3’UTR(图5E),其中m5C位点位于CG富集区中(图5F)。KEGG分析显示,NSUN2介导的m5C修饰基因在多种癌症相关信号通路中显著富集,如Rap1信号通路、铂类耐药性和细胞周期过程的调控等(图5G)。这些数据表明,NSUN2介导的GC转录本中的m5C修饰与RNA代谢和癌症发展密切相关。
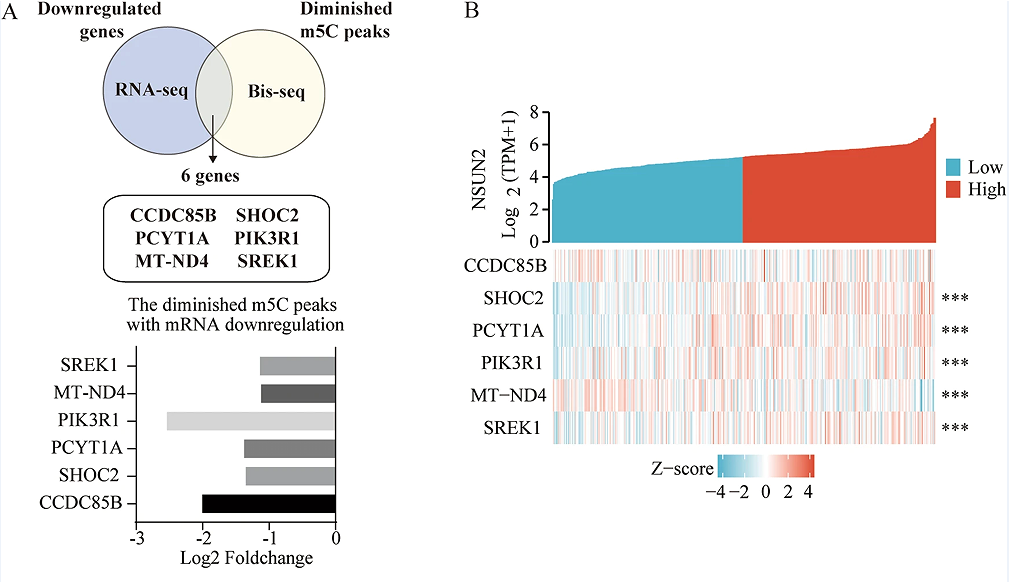
此外,作者还研究了m5C峰的变化是否会影响mRNA的表达水平。通过筛选了NSUN2-KO BGC- 823细胞中下调基因下降的m5C峰,鉴定了NSUN2的6个候选靶点,包括CCDC85B、SHOC2、PCYT1A、MT-ND4、SREK1和PIK3R1(图6A)。共表达热图显示,NSUN2的表达与除CCDC85B外的所有这些基因均密切相关(图6B)。之后qRT-PCR证实了NSUN2基因敲除后,PIK3R1和PCYT1A的表达水平显著降低,因此,作者推测PIK3R1和PCYT1A可能是NSUN2修饰的m5C的靶基因。综上,NSUN2介导的m5C修饰影响了GC的形成。
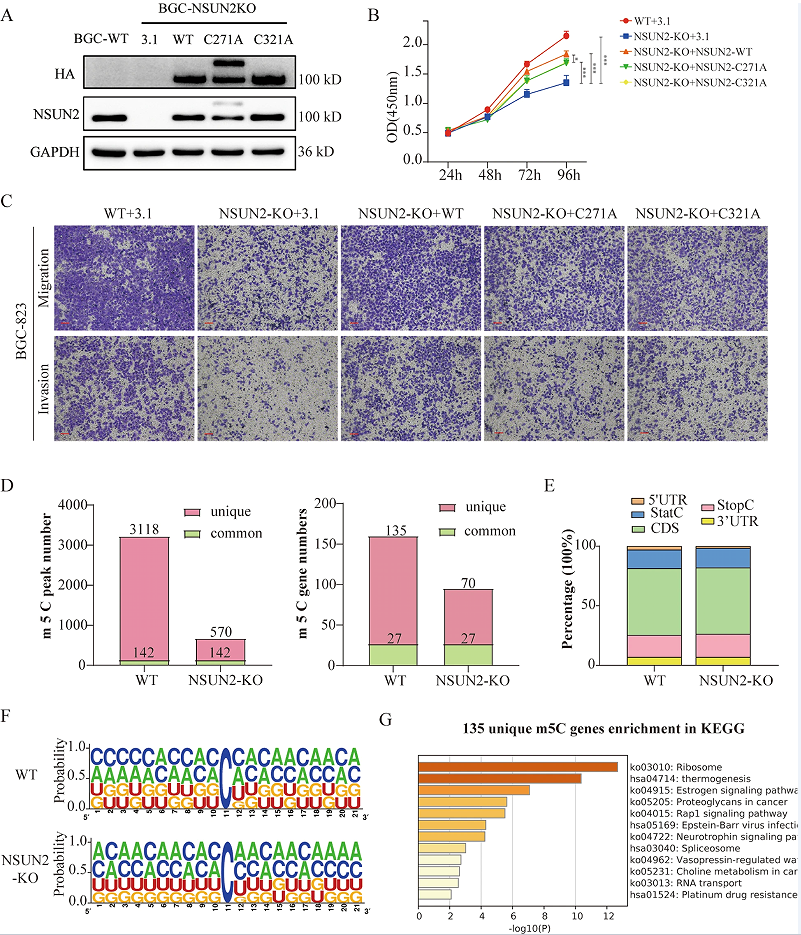
图5 NSUN2的致癌性部分依赖于m5C的修饰
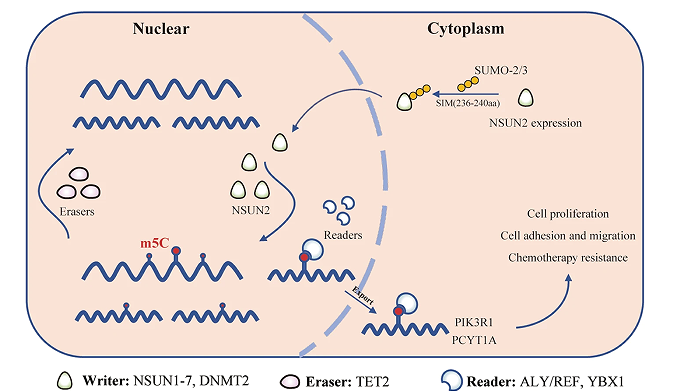
本研究通过m5C bis-seq分析,证实了NSUN2在胃癌中表达上调并与不良预后相关,并通过测定了m5C在GC mRNA中的分布,发现m5C修饰的基因主要参与了多种癌症相关的信号通路,SUMO化修饰-NSUN2-m5C轴可能是胃癌和泛癌治疗的一个新的诊断和治疗靶点(图7 )。
云序生物m5C甲基化研究五大模块
01 m5CRNA甲基化测序m5C RNA甲基化测序(m5C-seq)
对m5C RNA甲基化,目前最流行的检测手段为m5C-Seq技术,适用于m5C RNA甲基化谱研究,快速筛选m5C RNA甲基化靶基因。云序可提供mRNA和多种非编码RNA的m5C测序:
- m5C 全转录组测序(涵盖mRNA,LncRNA,circRNA)
- m5C LncRNA测序(涵盖LncRNA和mRNA)
- m5C Pri-miRNA测序(涵盖Pri-miRNA和mRNA)
- m5C mRNA测序
02 检测整体m5CRNA甲基化水平
LC-MS/MS检测整体RNA甲基化水平精准高效,可以实现一次检测,9类修饰水平检测,一步到位。
03 m5CRNA甲基化上游酶的筛选
m5C RNA甲基化相关酶PCR芯片
寻找上游直接调控m5C RNA甲基化的甲基转移酶。
04 m5CRNA甲基化靶基因验证
meRIP-qPCR
云序提供各类不同修饰的meRIP-qPCR服务,可针对mRNA,lncRNA,环状RNA等不同类型的RNA分子进行检测,低通量验证RNA修饰靶基因表达水平。
05 机制互作研究
优势一:云序累计支持客户发表 100 + 篇RNA修饰SCI论文,合计影响因子 960 +
优势二:累计完成数千例 RNA甲基化测序样本,覆盖医口、农口等各类样本。
优势三:可检测mRNA和各类非编码RNA(circRNA,lncRNA,Pri-miRNA等)。
优势四:提供m5C一站式服务:m5C整体水平检测、m5C-seq、MeRIP-qPCR验证、RIP和RNA pull-down等。
优势五:超微量MeRIP测序技术,RNA量低至500ng起。
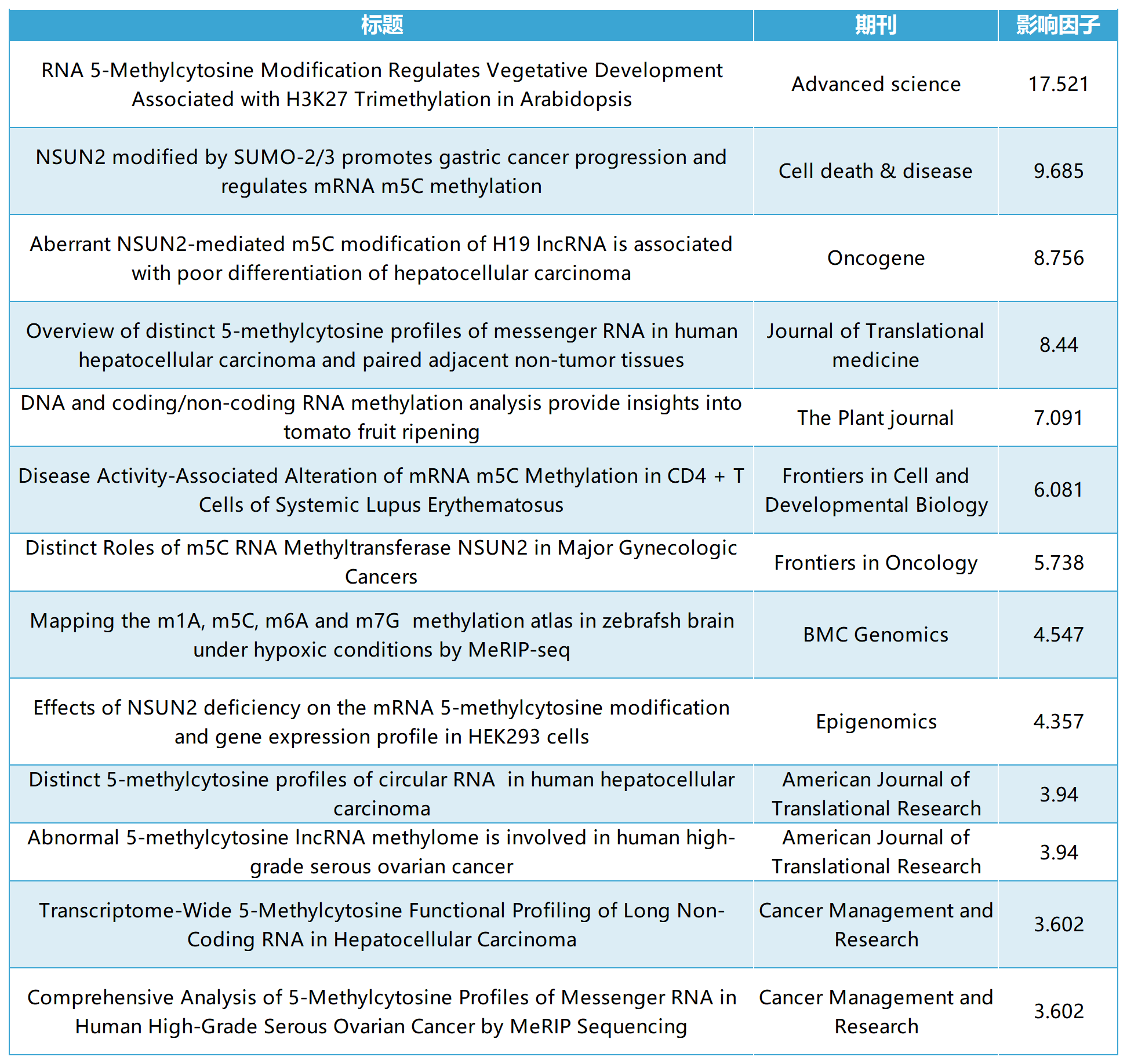
云序客户m5C修饰部分文章列表
往期回顾
上海云序生物科技有限公司 商家主页
地 址: 上海市松江区莘砖公路518号24号楼4楼
联系人: 戴小姐
电 话: 021-64878766
传 真: 021-64878766
Email:market@cloud-seq.com.cn;liuqingqing@cloud-seq.com.cn
